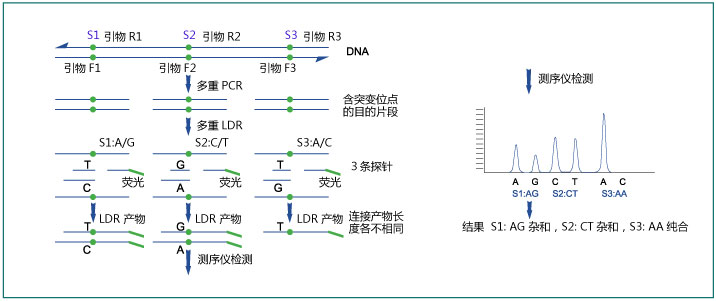
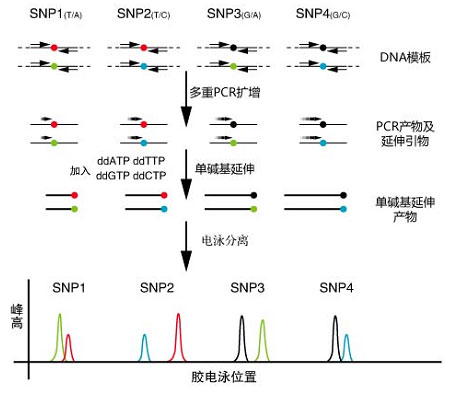
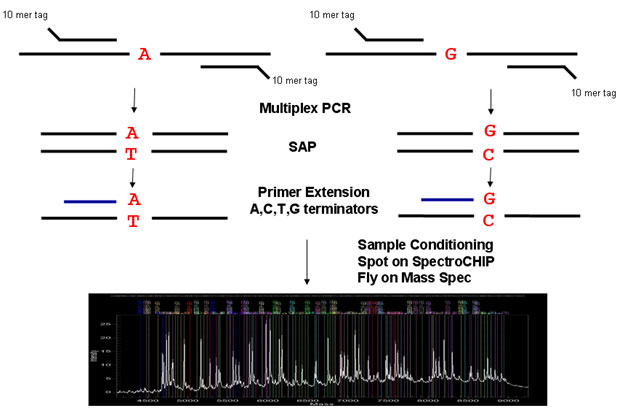
- 科研服务
- 定制产品
- 目录产品
- 细胞库
- MDA-MB-231-PR
- LOVO
- SK-N-BE(2)-C
- MDA-MB-231
- Daoy
- L6
- EL4
- SW1990
- SHI-1
- EL-4-B5
- H1581
- H1299
- RT112
- MKN28
- 3T3-L1
- MCF-10A
- mESC
- CV-1
- CAL-27
- DPCP2
- BGC-823
- SiHA
- Thp-1
- HCC827
- SH-SY5Y
- SNU368
- QSG 7701
- colo201
- DU145
- MONOMAC6
- MoLM13
- SK-HEP-1
- H22
- BaF3
- BRL
- H295R
- HEL
- Rat VSMC
- hepa1-6
- KU812

- 质粒库
- EOLA2
- RG9MTD2
- GPR68
- CDC45L
- ACOX3
- TMEM187
- ARD1A
- ZRSR2
- SYN3
- DGCR14
- CHAF1B
- ZNF239
- FZD4
- GAN
- PIP5K1B
- ALX1
- YEATS4
- MFAP5
- FGF23
- PTP4A2
- FOSL1
- CSRP3
- RASSF7
- COIL
- SDPR
- PEX3
- SLC43A1
- PPM1D
- RGS5
- RAE1
- HIRIP3
- SUPT3H
- TEAD2
- TPST1
- FZD7
- NCK2
- BTRC
- NR0B2
- STK24
- BFSP2
- STX7
- SRPX
- SPOP
- SLC25A11
- TUSC3
- NME5
- TPST2
- PCGF2
- SLC30A1
- ZNF232
- ZNF230
- ZNF213
- ZNF207
- ZNF200
- ZNF193
- RNF113A
- SHOC2
- TRIM26
- DNALI1
- ZNF136
- ZNF134
- MKRN3
- ZNF70
- ZBTB25
- ZSCAN21
- ZKSCAN1
- ZNF32
- ZNF23
- RNF112
- ARMET
- GCM1
- ZNF212
- TFPI2
- MTERF
- TFEB
- RDBP
- HSD17B8
- SLC39A7
- BAT4
- SLC30A4
- SLBP
- ZYX
- MAPKAPK3
- CXCR4
- IL1R2
- PAX8
- TUBA1A
- DAP3
- BSND
- PTP4A1
- SLC25A16
- DEK
- SUCLA2
- TMEM11
- NRP1
- LIN7A
- FGF17
- HESX1
- FGF18
- WDR22
- CCNK
- CRADD
- IL18RAP
- CFLAR
- TNFRSF10C
- NAPA
- SNAP23
- FADD
- RIPK2
- RAB11A
- CD164
- CDS2
- ENC1
- IL18R1
- SLC5A6
- WASF1
- SKAP2
- RAB7L1
- MBD4
- HERC3
- BCL10
- CPNE1
- CCNA1
- EIF2S2
- SOCS2
- EIF2B3
- GGH
- NAE1
- ZNF259
- CDC16
- VNN2
- LDB1
- ALDH1A2
- CDK5R1
- WISP2
- GPAA1
- EIF2B2
- MAPKAPK5
- TNFSF11
- STK16
- PRKRA
- PDXK
- YARS
- DENR
- DEGS1
- CDK10
- TNFSF12
- BHLHB2
- PPAP2C
- BLZF1
- CGGBP1
- PIR
- AGPS
- BARX2
- CAMK1
- CSDA
- GAS7
- ZNF20
- PIAS1
- EIF3G
- EED
- SNX4
- TRADD
- B3GALT2
- B3GALNT1
- B3GALT4
- B4GALT3
- S1PR4
- STX11
- RUVBL1
- EIF3H
- PPAP2A
- EIF3D
- DDX3Y
- AKR1C3
- RNASET2
- SKAP1
- HSD17B6
- RFXANK
- STC2
- IKBKG
- EIF3I
- TALDO1
- TECTB
- TCP1
- VPS72
- TCEA2
- TBX6
- TBL1X
- SERPINA7
- TBCE
- SURF2
- TARBP2
- TDO2
- MAP3K7
- TAF11
- TAF7
- TAGLN
- TADA2L
- SYPL1
- SYK
- SURF6
- TNFRSF1A
- TBCC
- TIMP2
- ZNF22
- TNFAIP1
- TNF
- TMPO
- TRAPPC10
- GPR137B
- TM4SF4
- NR2E1
- TK1
- TRGV3
- TIMP3
- TCTE3
- THRB
- THBD
- TGIF1
- LEFTY2
- TFDP2
- TFDP1
- TFAP4
- TERF1
- SULT2A1
- TIMP4
- SOAT1
- SRC
- SQLE
- SPP1
- SPN
- SPARC
- SOX15
- SOX10
- SOX5
- SURF4
- SOD2
- TRIM21
- SNRPN
- SNRPB2
- SNRPA1
- SNRPA
- FSCN1
- SNAPC3
- SNAPC2
- SNAPC1
- SNAP25
- SOX2
- STAR
- SULT1C2
- STYX
- STXBP3
- STXBP2
- STX4
- STX3
- SULT1A2
- STK11
- STK3
- SRD5A2
- STAU1
- AKR1D1
- ST13
- SSX2
- SSX1
- SSTR4
- SSTR2
- SSTR1
- SSR1
- TROVE2
- TNFRSF1B
- STC1
- VDR
- EIF4H
- WAS
- WARS
- VSNL1
- VRK1
- VIP
- VIM
- EZR
- UQCRC1
- VEGFB
- WNT2
- VDAC3
- VDAC2
- VDAC1
- VBP1
- VASP
- USF2
- USF1
- UROS
- TNFAIP6
- VEGFC
- XRCC2
- TFCP2
- ZNF3
- ZNF3
- ZIC3
- ZFP36
- YWHAE
- YWHAB
- YY1
- XRCC4
- LAT2
- XRCC3
- CORO2A
- XPA
- XG
- XBP1
- WNT2B
- WNT11
- WNT7B
- WNT7A
- WNT5A
- UPP1
- XRCC4
- TPT1
- TSSC1
- TSNAX
- TSHR
- TSC1
- TRIP6
- TRHR
- TRAF5
- TRAF2
- UQCRC2
- CRISP2
- TUBA4A
- TPMT
- TPM3
- TPI1
- TPH1
- TPD52L2
- TPD52
- TNNT2
- TNNI3
- TNNI1
- NR2C1
- UBE2E2
- UNG
- UMPS
- UGT2B10
- UGT2B7
- UGP2
- UCP2
- UCP1
- UCHL3
- UCHL1
- TSTA3
- UBE2G1
- DNAJC7
- UBB
- U2AF1
- TULP2
- TUFT1
- TUFT1
- TUFM
- TUBG1
- TUBB2A
- ZNF182
- UBE2H
- WDR68
- BCAS2
- SMNDC1
- OPRS1
- EFS
- NET1
- STUB1
- STAM2
- SPRY1
- FLOT1
- AP3S2
- BCKDK
- HNRNPR
- RASGRP2
- COQ7
- STX6
- GPA33
- ANGPTL7
- SSX3
- DDX39
- PARP3
- SPRY3
- KBTBD10
- CACNG3
- HMG20A
- HMG20B
- NPM3
- TFG
- PCGF3
- CLEC4M
- CNPY2
- TMEM5
- TRAIP
- AKR1A1
- DNAJA2
- IKZF1
- TNIP1
- NMUR1
- LANCL1
- RTN3
- APBB3
- PAK4
- BCKDK
- TRIM13
- COX4NB
- GPC6
- PPIF
- TSPAN1
- TSPAN3
- TSPAN5
- ACTR3
- ARPC1B
- ARPC3
- KCNK7
- DDX39
- PQBP1
- ACTR1A
- SNUPN
- RWDD2B
- IL18BP
- SCAMP3
- SCAMP2
- NR1H3
- SAE1
- AP1M2
- DNAJB6
- COL4A3BP
- ATP6AP2
- MPZL2
- DHRS2
- NME6
- PSME3
- ALG3
- RNF41
- LHFP
- SORBS3
- RCL1
- CTDSP2
- P2RY5
- PREB
- ABI2
- MBNL2
- EBI3
- G3BP1
- CLEC3A
- OPTN
- PDIA6
- ARL4A
- IRF9
- DHRS9
- TM9SF1
- COLEC10
- CCT4
- RABAC1
- OLFM4
- SLC35A1
- RPP38
- AGPAT1
- ARPC1A
- CACNG2
- PRDX4
- SCGN
- HBXIP
- ANP32B
- GLRX3
- RNASEH2A
- SSSCA1
- APPBP2
- SEMA4F
- STK25
- VAT1
- ARL6IP5
- MRCL3
- TFAM
- CHRNA6
- SPINT2
- YKT6
- MTX2
- SLMO1
- TUSC4
- LEFTY1
- RGS14
- MAB21L2
- RASL10A
- DRAP1
- POLR3C
- STAMBP
- ST6GALNAC2
- SC65
- PAICS
- CDC42EP3
- AHSA1
- DNPH1
- CREB3
- RAD51AP1
- GNB2L1
- SYNCRIP
- TMEM147
- ARIH2
- PGRMC2
- PMVK
- CDIPT
- UBAC1
- CD2BP2
- TINP1
- CDC42EP2
- ST3GAL6
- EMG1
- NDRG1
- ANAPC10
- CEPT1
- BTN2A2
- BTN2A2
- BTN3A3
- TUBB2C
- TUBB3
- SPRY2
- NDC80
- PECI
- TUBA1B
- CAP1
- NXF1
- HOXB13
- EIF3M
- SLC25A17
- UBE2E3
- TADA3L
- TIMM44
- LYPLA1
- HAX1
- VTI1B
- MAP3K7IP1
- TOMM40
- PPIE
- ACAA2
- FAM3C
- MCRS1
- N4BP2L2
- TIMM17A
- IFI30
- MAD2L2
- NOG
- BCL7B
- BCL7C
- CYTH2
- PDLIM7
- PDLIM7
- SCYE1
- DHRS3
- GCM2
- TBPL1
- MSC
- SOCS6
- PNMA1
- TBRG4
- IL32
- LRAT
- VAPB
- FAIM3
- AURKB
- BMP15
- LRRFIP2
- GCNT3
- ADIPOQ
- GDF15
- RPH3AL
- MYOT
- PIGL
- MED20
- ATG5
- THEMIS2
- EIF4E2
- IL27RA
- GPR55
- TM9SF2
- GPR52
- RAB9A
- RAB33A
- CNOT8
- B4GALT5
- TRIP13
- COPS2
- PTER
- CD83
- KCNAB3
- CIAO1
- CH25H
- ZMYM3
- CLDN12
- ANGPTL1
- SYT7
- PIAS2
- PAPSS2
- SLC7A6
- PSTPIP1
- UBE2M
- CLDN8
- RHBDL1
- CLDN2
- CLIC3
- SOCS3
- MPZL1
- SLC25A14
- NOL3
- SELENBP1
- STBD1
- WASL
- P4HA2
- CCRL2
- BCL2L11
- AIFM1
- SLC16A7
- DEDD
- BUB3
- RASSF9
- AP4M1
- LPAR2
- EBAG9
- SYNGR2
- CLDN10
- CCNE2
- SLC33A1
- PRPF4
- SF3B4
- SLC16A3
- SLC16A6
- SEC22C
- ATP6V0D1
- USP14
- UNC119
- DIRAS3
- SYNGR3
- RASSF2
- ZNF432
- NUPL1
- KEAP1
- KIAA0494
- IHPK1
- DAZAP2
- MRPL19
- PTDSS1
- SNX17
- RGP1
- EIF4A3
- TRIM14
- KIAA0247
- MLEC
- SNPH
- LAPTM4A
- NOS1AP
- HERPUD1
- TRAM2
- TTC35
- SLC25A44
- RIMS3
- OXSR1
- PDLIM1
- EEF1E1
- GNPDA1
- PDCD6
- ACOT8
- SCO2
- DGCR2
- ROD1
- REC8
- SPATA2
- USP3
- PSMD6
- ENDOGL1
- RBM8A
- JOSD1
- LPGAT1
- FAM20B
- SLC35E2
- SETDB1
- FARP2
- EPM2AIP1
- FGF19
- MED7
- ZNF254
- POLR1C
- BAG2
- BAG3
- TMEM59
- VPS4B
- HOMER1
- HOMER2
- GRAP2
- TP53I3
- QKI
- GGPS1
- KCNK6
- HAND1
- FAM189A2
- SNRNP40
- TMPRSS11D
- MTFR1
- KLHL21
- GPSN2
- SCAMP1
- ITM2A
- IER2
- IKBKE
- MORF4L2
- FEZ1
- GNA14
- GSTO1
- RAB3D
- TRAF4
- RNF14
- CYTIP
- KLK4
- WTAP
- PRDX6
- APBA3
- RBM39
- BRE
- SPAG7
- NR1D1
- GOSR2
- PDIA4
- MINPP1
- 案例分享
- 关于诺百
- 招贤纳士